Transparisation des tissus
La transparisation des tissus est une technique récente qui permet de rendre les organes optiquement transparents afin de faciliter leur observation au microscope. Elle permet de visualiser un environnement complexe (eg cerveau) dans son intégralité sans recourir à des coupes sériées.
Méthode Clarity

Principe
Étape 1 : Perfusion et post-fixation des échantillons
Etape 2 : Imprégnation et polymérisation dans un hydrogel
Étape 3 : Transparisation qui va permettre l’élimination des lipides et homogénéiser l’indice de réfraction de l’échantillon.
Etape 4 : Réalisation de l’immunomarquage si besoin
Avantages de cette méthode
- Immunomarquage facilité car réalisé après l’élimination des lipides
- Rapidité
- Préservation des signaux fluorescents endogènes (YFP, GFP, …)
Equipement disponible sur la plateforme :
Set Up X Clarity (Logos Biosystems)
Méthode Disco

Principe
Etape 1 : Blanchiement et perméabilisation des échantillons
Etape 2 : Immunomarquage
Etape 3 : Transparisation chimique permettant l’élimination des lipides
Etape 4 : Imprégnation dans le milieu de montage
Avantages de la méthode :
Préservation de la morphologie et de la taille des échantillons
Meilleure résolution à l’acquisition des images
Microdissection Laser

Préparation des échantillons
La préparation des échantillons est une étape fondamentale avant la microdissection. Il faut prendre en compte les contraintes liées à l’analyse en aval (ex. quantification d’ARNm, dosage de protéine etc.).
En général, les tissus frais et congelés sont préférés pour isoler l’ARN et les protéines. Les coupes congelées peuvent être obtenues rapidement sur un cryostat. Un ARN de haute qualité est obtenu à partir de tissus congelés immédiatement après la chirurgie, ce qui minimise sa dégradation au fil du temps par les RNases (activité tissu dépendant).
L’ADN, L’ARN peuvent aussi être préparés à partir de matériel congelé ou fixé au formol, même après enrobage paraffine. L’avantage de travailler avec des coupes enrobées de paraffine est une qualité morphologique supérieure par rapport aux coupes congelées. La préservation de la morphologie peut être critique pour le diagnostic et pour la sélection finale de cellules spécifiques à microdisséquer. Cependant, la préparation d’ARN à partir de coupes de paraffine fixées peut être difficile en raison de la dégradation de l’ARN par le formol (fixateur généralement utilisé pour les inclusions en paraffine).
Apres microdissection, les échantillons sont récupérés dans des tubes et l’extraction (ARN, ADN, Protéine) réalisée rapidement.
Equipements disponibles sur la plateforme :
1 LMD7000 (Leica) lien vers page équipement
Multiplex
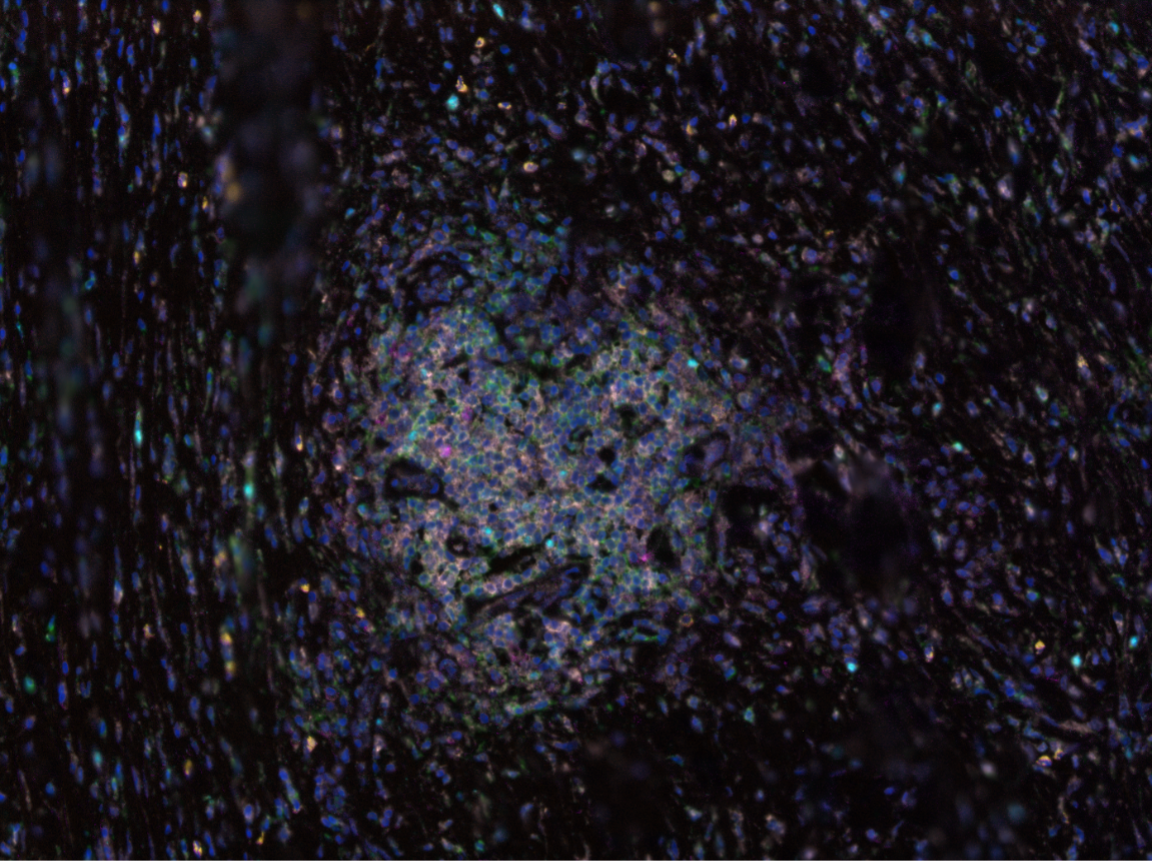
L’immunohistochimie en multiplex est une technique plus récente qui permet de réaliser de multiples immunomarquages séquentiellement et donc d’étudier de nombreux co-marquages.
Principe de l’immunohistochimie en multiplex
Elle est basée sur le système d’amplification de signal par la Tyramide.
Méthode de l’immunohistochimie en multiplex
- Le premier immunomarquage est réalisé sur la coupe
- La peroxydase conjuguée à l’anticorps secondaire catalyse la conversion des molécules de tyramide en radicaux libres hautement réactifs qui vont former des liaisons covalentes à proximité de l’épitope
- L’élimination du couple anticorps primaires/anticorps secondaires par chauffage (« stripping ») préserve la TSA liée et le fluorochrome rapporteur
- Le processus peut être répété avec un nouvel anticorps primaire, jusqu’à ce que toutes les cibles d’intérêt soient détectées, en utilisant un fluorochrome de longueur d’onde différente pour chaque marquage.
La technologie repose sur la capacité :
- De la TSA couplée au fluorochrome à être liée de manière covalente aux tissus
- À éliminer efficacement les anticorps entre les cycles de coloration
- Des antigènes à résister à la chaleur
Avantages de la technologie de l’immunohistochimie en multiplex
- Signal augmenté (x10 à x100)
- Résolution exceptionnelle
- Volume d’anticorps primaires utilisé réduit
- Diminution du temps d’incubation (min 1h)
- La liaison covalente établit par la technique est idéal pour le multiplexage
A disposition sur la plateforme Histomics de l’Institut du Cerveau (Paris)
La plateforme met à disposition un ensemble de réactifs pour la réalisation de vos immunohistochimies (ex : sérum, BSA, anticorps secondaires fluorescents, système de révélation, DAB, Dapi, milieu de montage, …etc).
Kit Akoya :7 fluorophores : Opal480, Opal520, Opal 540, Opal 570, Opal 620, Opal 650 et Opal 690
Les anticorps primaires restent à la charge des utilisateurs.
|
|
|
Hybridation in situ
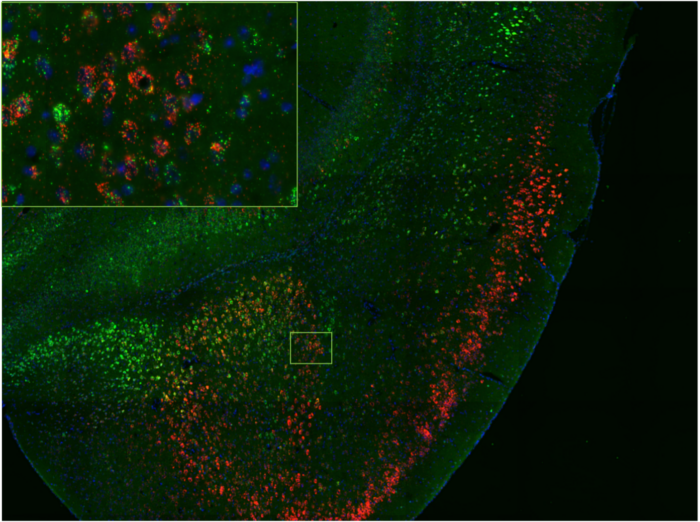
Cerveau murin en coupe congelée
Transcriptomique spatiale
GeoMx Digital Spatial Profiler
|
|
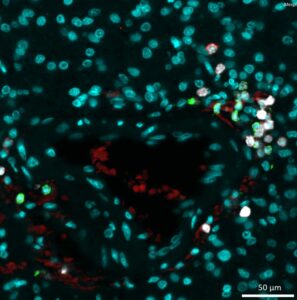
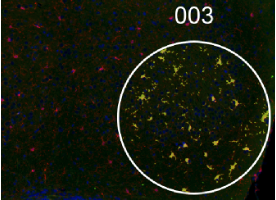 Cerveau de souris. IBA-1 (rouge) et noyau (bleu). Profilage de populaire de microglies (zones jaune). |
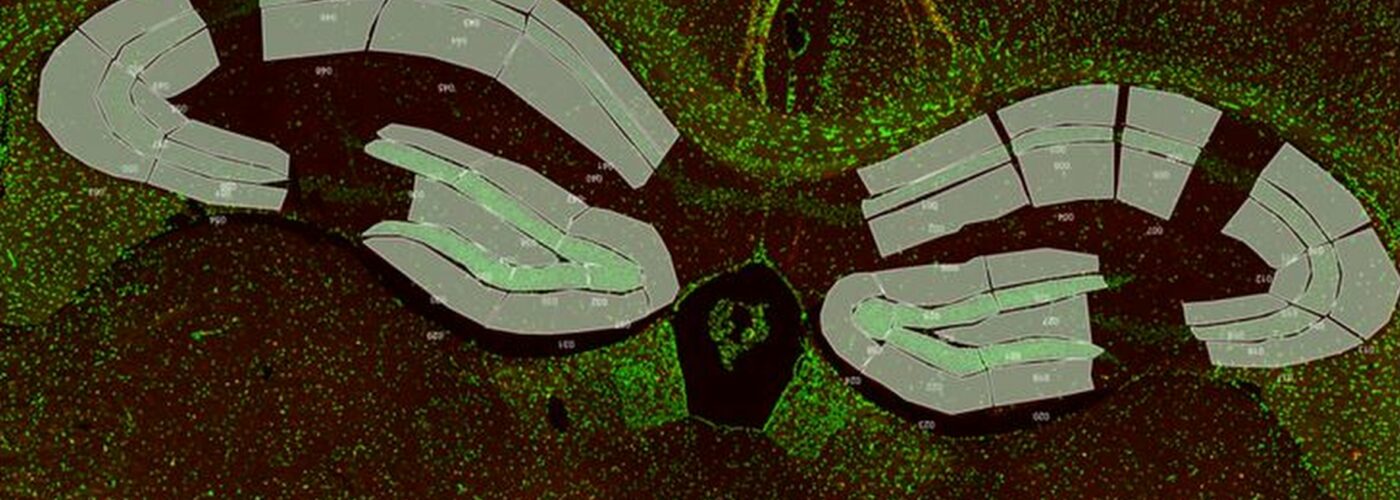
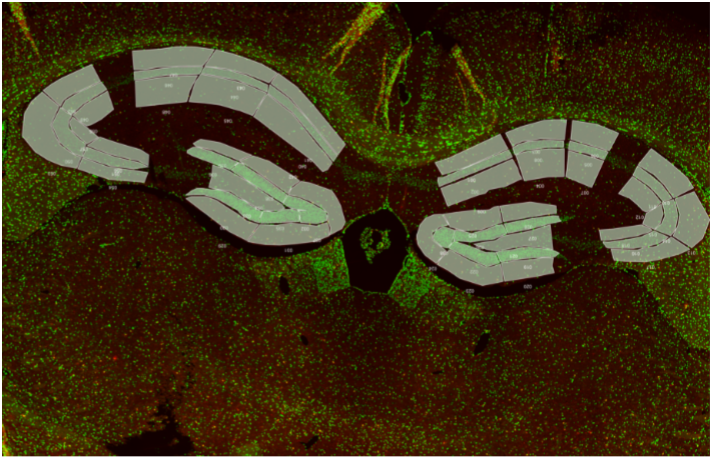
La technologie GeoMx DSP est une technologie de transcriptomique et protéomique spatiale basée sur le séquençage nouvelle génération. Elle permet d’étudier plus de 20 000 ARN et 150 protéines. Elle est adaptée aux divers tissus (congelé ou paraffine). Les ARN ou protéines d ’intérêts sont détectés par des sondes ou des anticorps couplés à une molécule photoclivable avec un code-barre spatial permettant ainsi de les localiser dans le tissu. Nous fournissons un panel de marqueurs morphologiques (IBA-1, GFAP, Olig2, CD3, PanCK…) permettant de sélectionner des populations cellulaires ou des régions d’interet dans le tissu. La lame histologique est imagée sur la plateforme GeoMx DSP. A l’aide de marqueurs morphologiques, les régions d’interet sur la coupe de tissu sont sélectionnés puis illuminées par la lumière UV afin de cliver les oligonucléotides contenant les code-barres spatiaux. Ces oligonuclétides sont aspirés et collectés dans une plaque à 96 puits. Ils sont ensuite amplifiés et séquencés sur une plateforme illumina. GeoMx DSP intègre ensuite les données de séquençage aux données histologiques pour une analyse de transcriptomes ou protéomes.
Visium
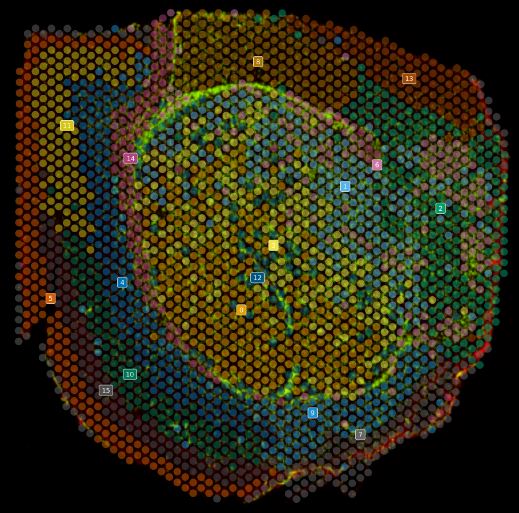
Cartographie de clusters cellulaires sur une coupe de cerveau de souris greffé de cellules de glioblastome humain.
La technologie Visium est une technologie de transcriptomique spatiale basée sur le séquençage nouvelle génération permettant une analyse de l’intégralité de transcriptomes sur une coupe de tissu. Le Visium utilise une lame recouverte de spots greffés de poly(dT) permettant ainsi de capturer des ARNm du tissu. Une coloration HE ou une immunofluorescence est réalisée afin d’obtenir des informations morphologiques du tissu. Le tissu est ensuite perméabilisé pour libérer des ARNm qui seront capturés par des poly(dT) sur la lame Visium. Les ARNm capturés sont rétro-trancrits en ADNc qui sont ensuite amplifiés et séquencés sur une plateforme illumina. Les données de séquençage et l’image histologique sont alignées dans le logiciel SpaceRager de 10X Genomics permettant ainsi de cartographier l’expression de gènes sur la coupe de tissu.